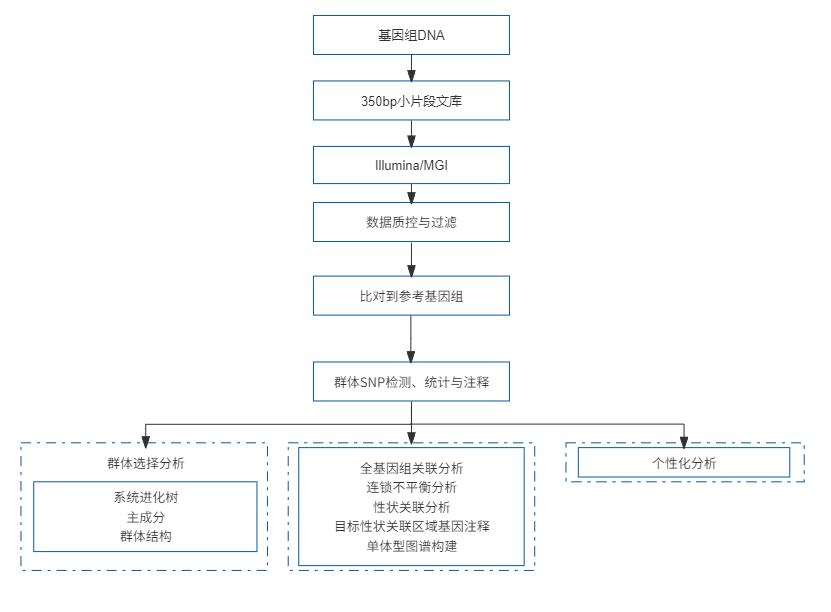
全基因组关联分析 (Genome-Wide Association Study,GWAS)在动植物的研究中对于重要性状特别是复杂性状的定位有着快速、准确的优点。基于高通量测序对某种农作物或禽畜的代表性品种、地方种或野生种进行基因分型,结合准确的表型数据可对农作物或禽畜重要复杂性性状进行定位。特别是在包含了野生种、驯化种和改良种的群体中,结合群体进化分析对收到驯化和改良的重要基因进行定位,是研究作物或禽畜微进化及驯化改良表型的重要思路。
全基因组关联分析快速定位影响水稻农艺性状的新基因
Genome-wide association study using whole-genome sequencing rapidly identifies new genes influencing agronomic traits in rice
期 刊:Nature Genetics 发表时间:2016.06 发表单位:日本名古屋大学
影响因子:29.352
对于鉴定未知基因与 QTL 的关联存在较大困难的原因在于:1、具有丰富多态性的作物群体材料常常存在强烈的群体结构效应,进而导致表型与分子标记之间的假阳性关联结果。2、LD 衰减距离过长导致未知基因鉴定困难,特别是当单个LD 衰减范围内存在多个候选基因表现出显著信号时,对于与目标基因关联的真实基因需要通过额外的实验进行确认。
材料:选取了没有较高群体结构、彼此之间有一定血缘关系的176日本粳稻品种,同时群体又表现出很高的群体多态性
建库:DNA小片段文库
测序:Illumina HiSeq2000,PE100,5.8X
分析:群体结构分析,LD分析,关联分析
群体表型鉴定
选择表型呈正太分布且组间存在丰富的差异的材料;基于样本间SNPs多态性的群体结构分析则表明在这些表型差异丰富的材料之间不存在明显的群体分层。群体的 LD 衰减物理距离在445kb(r2 = 0.2),与已报道的具有更高表型多态性的群体相比,LD 衰减比较一致,说明材料的选择对控制 LD 衰减有一定的帮助。
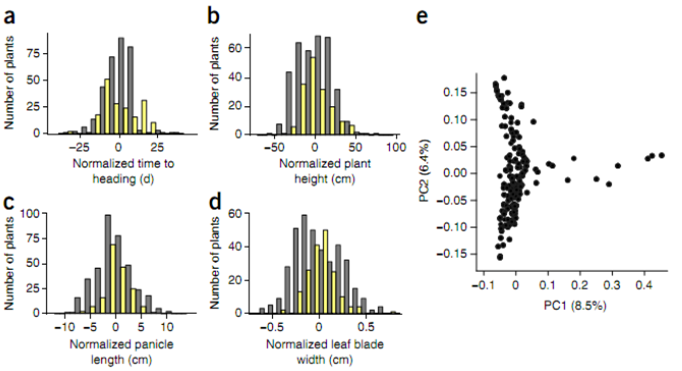
图1 群体表型数据统计与主成分分析
GWAS快速鉴定性状关联基因及验证
文章通过使用混合线性模型进行 GWAS 关联分析,鉴定到了26 个LOD 值大于4.77 的位点,其中有 3个最高的信号位点分别位于染色体 1,6,11,两个峰点的位置与已报道的抽穗期相关基因Hd6 和Hd2的QTL 定位结果一致,分别定位与染色体3 和染色体7。对于GWAS 定位结果的验证,采用已报道的 QTL 来验证是最具说服力的。
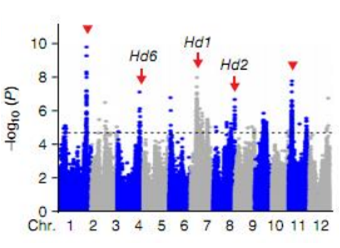
图2 与水稻拔节相关的QTL定位结果
新基因的鉴定
对于未经报道的QTL定位信号,其中位于染色体 1 的候选区域被锚定在36.30Mb 到36.65Mb之间(346kb),该候选区域包括了91个与抽穗期显著关联的位点和7个基因,其中基因LOC_Os01g62780与拟南芥的HESO1基因同源,该基因在拟南芥中表现为延迟开花。同样,位于第11号染色体上也挖掘到了与水稻抽穗期相关的候选基因LOC_Os11g08410。研究者也对水稻的分蘖数、叶宽等性状进行了全基因组关联分析,获得了许多控制上述农艺性状的候选基因。
为保证GWAS 分析结果的准确性,文章在材料选择方面选择了群体结构分层不明显的水稻材料。基于基因的关联分析对农艺性状 GWAS 分析检测的假阳性结果有很好控制效果。
Yano K, Yamamoto E, Aya K, et al. Genome-wide association study using whole-genome sequencing rapidly identifies new genes influencing agronomic traits in rice[J]. Nature Genetics, 2016.